使用PyMOL绘制蛋白与配体分子结合模式图
摘要
本文将带大家使用免费开源的PyMOL软件绘制D715-2441蛋白与与PB2cap配体分子的结合模式图。
我们先来看一张成品图: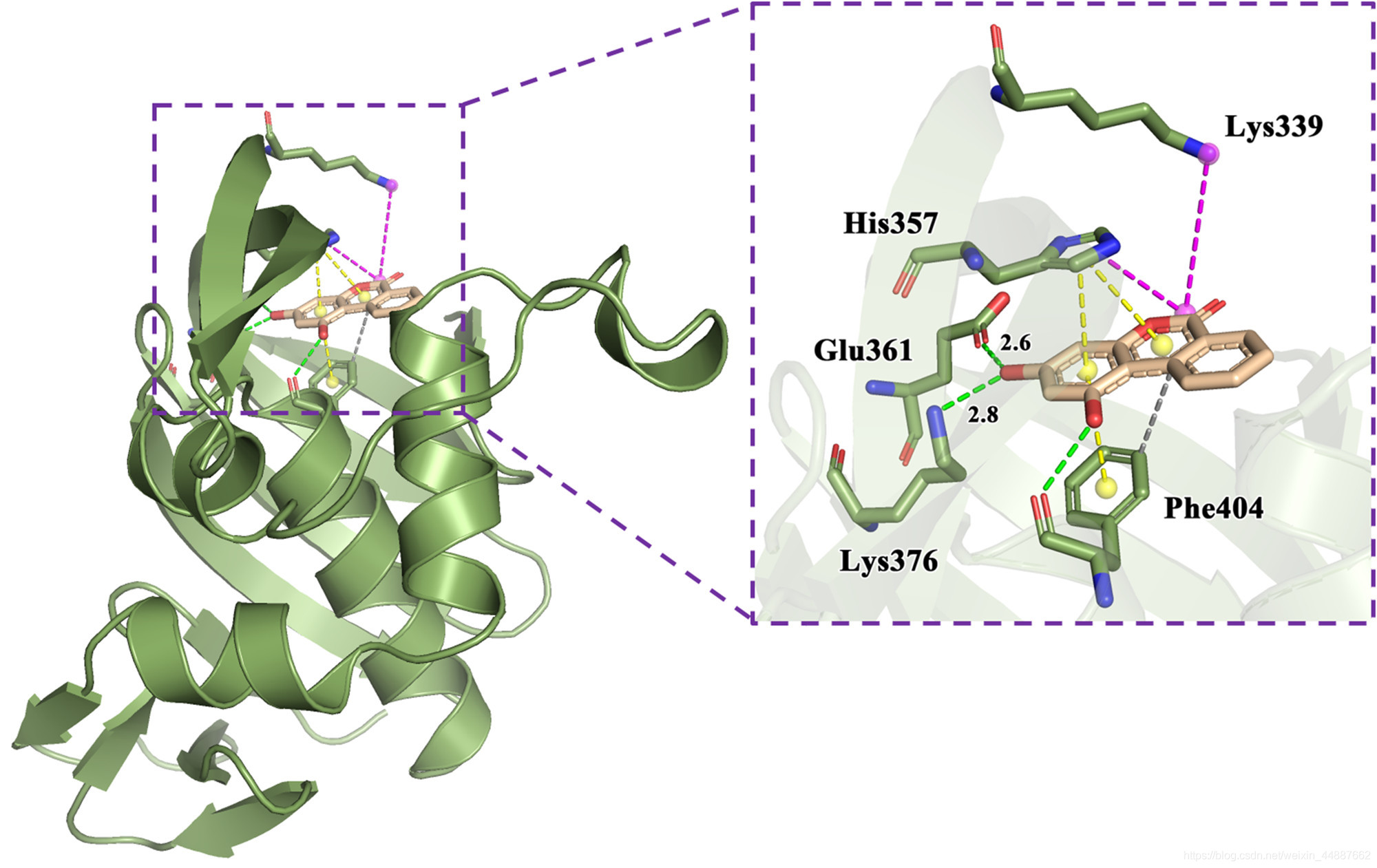
研究背景
根据分析结果,我们获得以下相互作用:
1、化合物D715-2441的芳环夹在氨基酸残基H357和F404之间并形成π-π堆积作用,还与F404形成疏水作用;
2、化合物苯环上的羟基与E361的羧基和K376的N+原子形成氢键作用,另一个羟基与F404的O原子形成氢键作用;
3、化合物吡喃酮环酯基与K339的N+原子和H357的咪唑基形成盐桥作用。
操作步骤
PyMOL作图通常是对特定对象进行展示形式、颜色、字体等方面的修饰。PyMOL的精髓在于命令行与鼠标的配合使用,本文高度依赖命令行,请做好心理准备。大多数命令格式较为一致,请注意总结归纳。
**1 设定工作目录,打开文件 **
打开PyMOL软件,导入刚才下载的复合物PDB文件(rli-D715-2441-1.pdb)。
注意:有两种方法导入文件,一种是鼠标点击菜单栏File -> Open…,另一种是通过命令行。在这里,我们采用命令来操作,因为这样可以同时把工作目录设置好,方便后续保存文件。
假设我们的工作目录为D:\demo。注意:路径中不要有中文和空格,这是导致很多计算类软件(尤其是免费软件)出现问题的原因。
在PyMOL上界面或下界面的命令输入框输入以下命令(PyMOL>表示这是PyMOL的命令),每行输入完毕后按【Enter】键:
2 设置操作对象
结合模式图通常涉及到受体(蛋白)与配体(小分子化合物)两部分(有的还可能涉及水分子、金属离子、辅酶;等等),需为之分别设置对象,以便后续操作。设置配体对象:按照下图顺序,将配体设置为lig对象。
3 设置关键残基对象
关键残基包括:A链的K339、H357、E361、K376和F404。由于该蛋白只有一条链,在使用命令选择残基时,可以不用指定链名。按照下图顺序,将关键残基设置为res对象。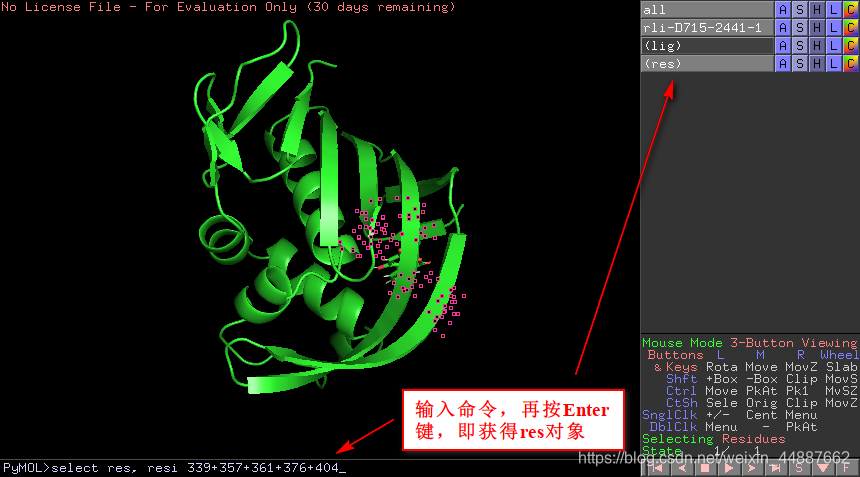
**4 显示所需,隐藏多余 **
在PyMOL中输入以下命令: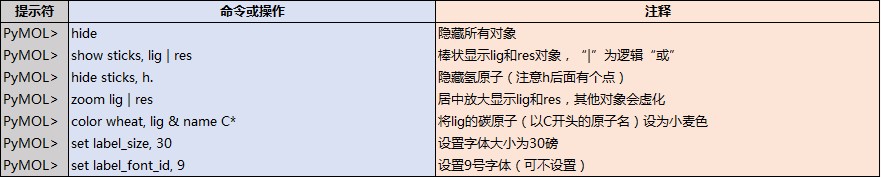
然后,进行鼠标操作: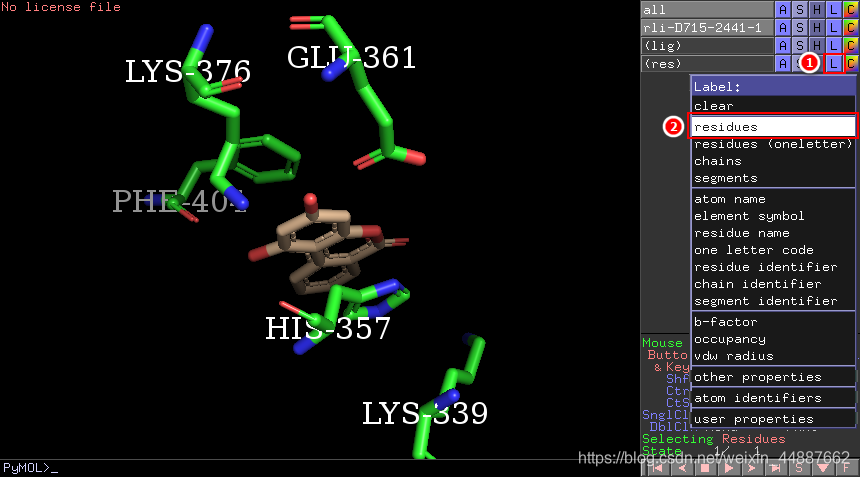
**5 绘制相互作用力 **
前面说过,命令行和鼠标的配合是很重要的。这里再简单讲讲鼠标的几个操作用法:
A) 拖动鼠标左键:按住右键,任意方向拖动鼠标,使视图转动;
B) 上下拖动鼠标右键:按住右键,上下拖动鼠标,使视图缩放;
C) 拖动鼠标中间滚轮:按住滚轮,任意方向拖动鼠标,使视图移动;
D)滚动鼠标中间滚轮:拉进或拉远镜头,使近处原子逐渐显示或隐藏。
我们参照平台给出的结合模式图(本文第二张图),通过下面的键鼠操作来绘制相互作用力。
· 绘制π-π堆积与盐桥作用
首先,确保鼠标模式(Mouse Mode)为Viewing状态,选择(Selecting)模式为Atoms状态(位置在PyMOL右下方)。然后,按照下面步骤操作: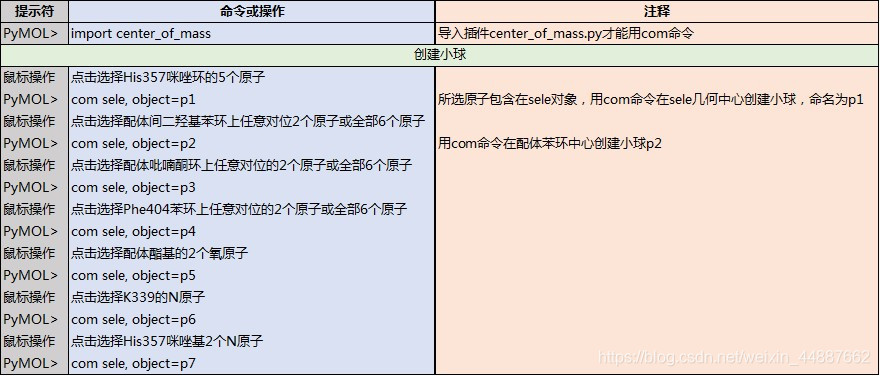 然后,点击PyMOL菜单栏的Wizard -> Measurement,然后根据示意图点击刚才创建的小球,两两一组,便画出虚线(相应地,生成对象measure01~05)。
然后,点击PyMOL菜单栏的Wizard -> Measurement,然后根据示意图点击刚才创建的小球,两两一组,便画出虚线(相应地,生成对象measure01~05)。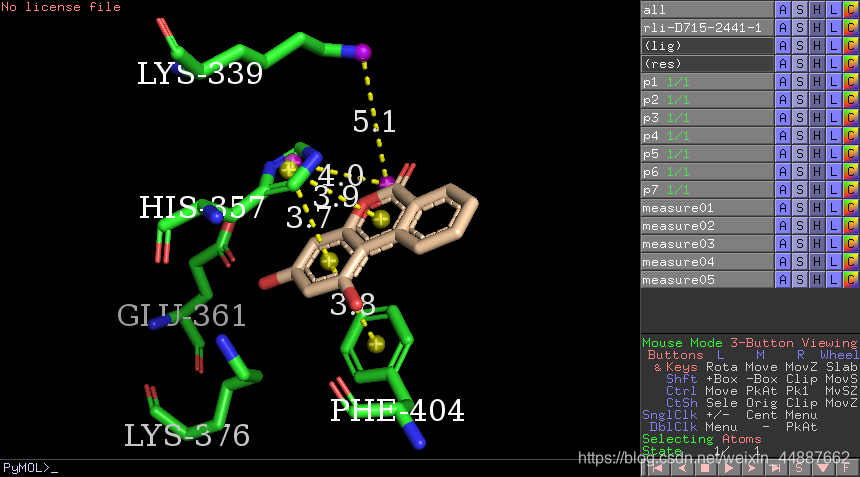
· 绘制其他作用力
采用相同的办法,把氢键和疏水作用也画出来。由于这些作用力的两端刚好落在原子上,因此,不用绘制小球。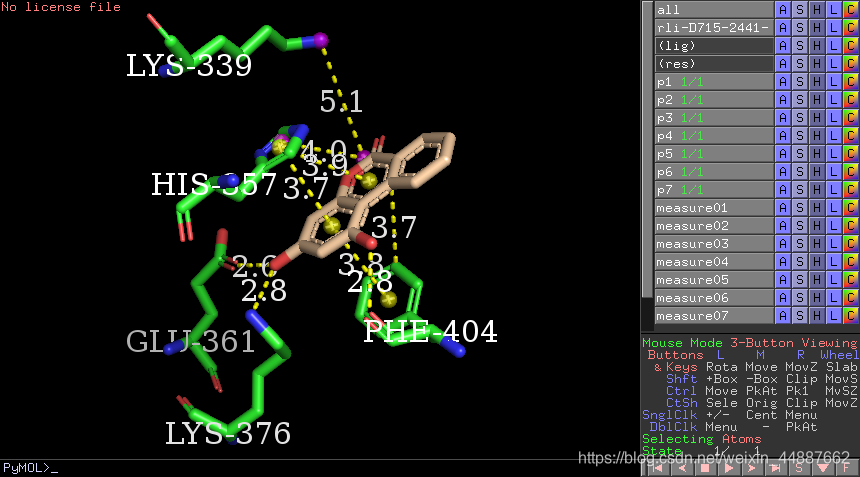
**6 最后加工 **
为方便调整,也为图片规格符合要求(通常,出版社要求全版图片宽度≤2000像素,半版图片宽度≤1000像素,分辨率≥300
dpi),我们将PyMOL界面放大或靠边最大化,并将上半部分的对话框往上拉到最小,让视图最大。然后放大、摆动视图至合适的角度,使尽可能清晰地看到各关键细节。
基本的创建工作已经完成,接下来进行细致的修饰。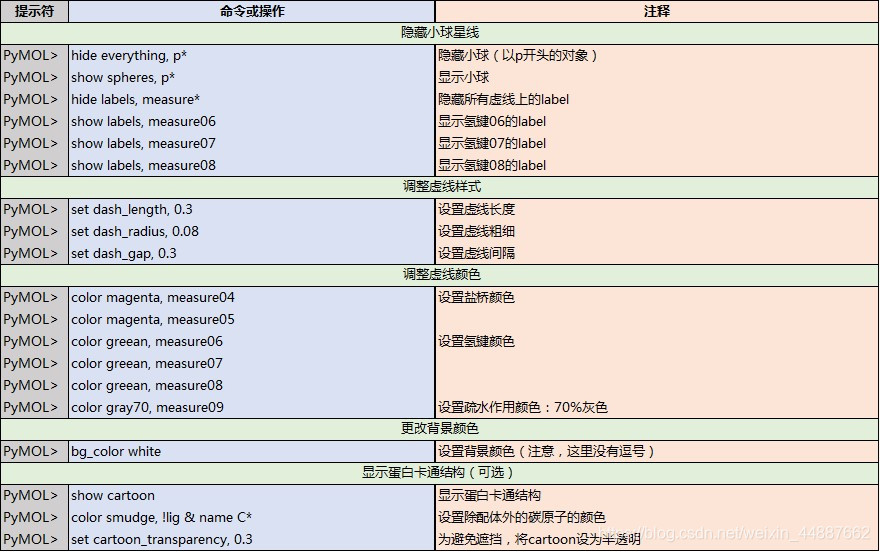 我们还可按下图步骤微调cartoon样式:
我们还可按下图步骤微调cartoon样式: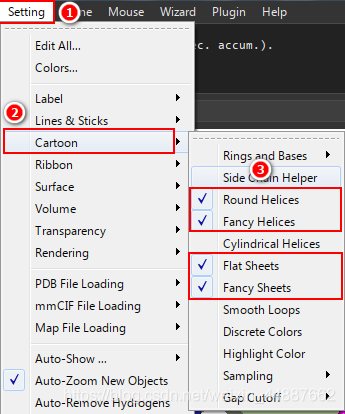
还可移动label的位置(设置Mouse Mode为Editing模式后,按住Ctrl键,用鼠标左键或右键拖动label;必要时,可先将cartoo隐藏起来,等调整好label后,再显示出来),让视图更清晰。经过上述一系列操作,得到下图: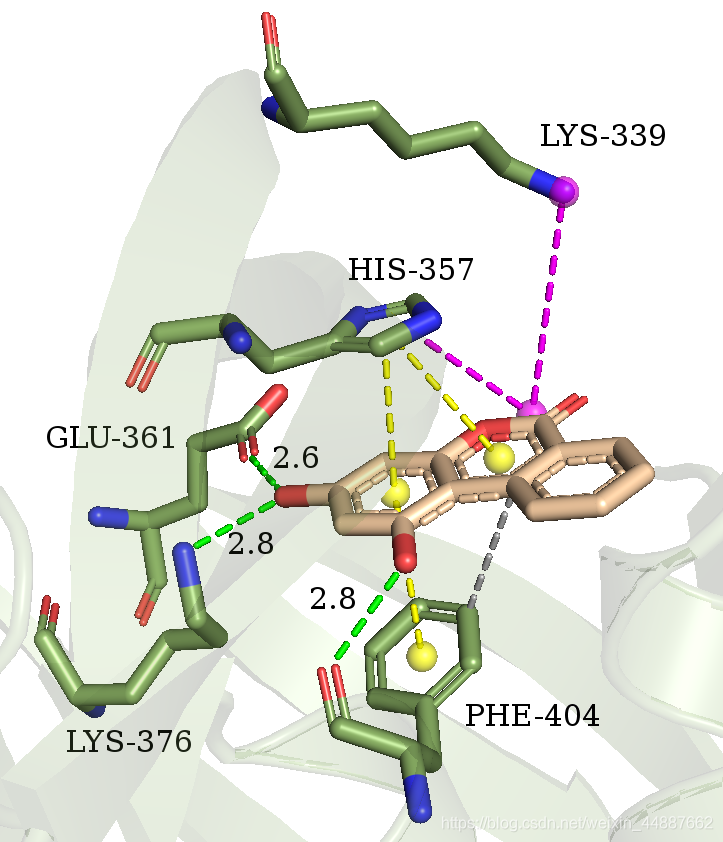
**7 生成图片 **
上面的图已经可以发表文章了,但为了更好看,还需要进行一步:光线追踪(Ray tracing)。
继续使用命令: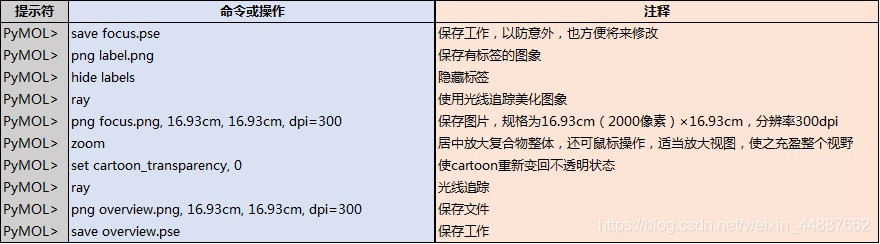
至此,我们拿到3张图片:focus.png、label.png和overview.png。
说明
本文摘编自高质量PyMOL作图教程,遵循 CC 4.0 BY-SA 版权协议。